본 글에서는 U-Net의 구조를 상세히 다루고 있으며, U-Net++에 대해서도 간단히 다루고 있다.
본 글은 U-Net 논문과 기타 기술블로그를 참고하여 작성한 글이다.
다음은 U-Net 논문의 링크이다.
U-Net: Convolutional Networks for Biomedical Image Segmentation
There is large consent that successful training of deep networks requires many thousand annotated training samples. In this paper, we present a network and training strategy that relies on the strong use of data augmentation to use the available annotated
arxiv.org
U-Net은 이미지 세그멘테이션(image segmentation)을 위한 모델이다. ISBI cell tracking challenge 2015 대회에서 등장하였으며, 세포 이미지에 대한 뛰어난 세그멘테이션 성능을 보이며 우승을 차지하였다. 해당 대회에서 제공한 학습 데이터 이미지는 총 30장 밖에 되지 않았음에도 U-Net는 뛰어난 성능을 보였다. 적은 학습 데이터로도 뛰어난 성능을 보인 U-Net의 구조와 Data Augmentation 기법에 대해 살펴보자.
글의 순서는 다음과 같이 진행된다.
- Introduction
- U-Net의 Architecture (Contraction Path + Expansive Path)
- 입력 이미지에 적용한 기법들 (Overlap tile & Mirroring the input image)
- Data Augmentation
- Training & Experiments
- Conclusion
- U-Net++
1. Introduction
 |
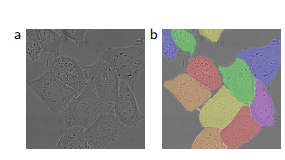 |
전형적인 CNN 모델은 이미지를 입력하면, 이미지 하나 당 하나의 class label이 출력되는 'classification' 작업을 수행한다. 그런데 실제로 사용할 때에는 localization을 포함한 Classification이 필요한 경우가 많다. 이는 이미지 전체가 아닌 이미지 내부의 각 픽셀에 해당하는 labeling이 필요함을 의미한다. 특히 Biomedical image에는 한 이미지 안에 여러개의 세포가 들어있어 더욱 세밀한 작업이 필요하다.

그러나 Biomedical 분야에서는 대량의 training set를 확보하기 어렵다. 그동안 training set 확보가 어려울 때에는 주로 Sliding window 방식을 통해 문제를 보완해왔다. Sliding window 방식이란 이미지를 윈도우 사이즈에 맞추어 patch 단위로 입력값으로 사용하는 방식이다. 하나의 이미지 당 여러 개의 패치가 만들어지므로 풍부한 데이터셋을 구현할 수 있다.
그러나 기존 sliding window 방식은 이전 window에서 검증이 끝난 patch 구역을 다음 sliding window에서 다시 검증해야 하므로 연산에 있어서 효율적이지 않았다. 이에 U-net에서는 이미 검증이 끝난 부분은 건너뛰고 다음 patch부분부터 검증하여 연산 속도를 빠르게 했다.
그럼에도 본질적으로는 patch 단위의 입력을 사용하는 것이기 때문에 localization accuracy와 use of context 간에 trade-off가 발생하는 문제 - 즉 전체를 보는 것과 부분을 보는 것 사이의 trade-off는 남아있다.
U-Net에서는 이러한 문제점들을 해결하기 위해 U 모양의 "fully convolutional network"를 제안했다.
2. U-Net의 Architecture
그럼 이어서 U-Net의 구조를 살펴보자. U-Net은 크게 Contraction Path와 Expansive Path로 구성되어 있다. 두 Path가 어느 정도 대칭적이어서 U자형 아키텍처를 가진다.
- Contraction Path(축소 경로) : receptive field를 키우면서 입력 이미지의 주요 특징 추출
- Expansive Path(확장 경로) : Localization을 위해 feature map의 해상도를 키움
1. Contraction Path
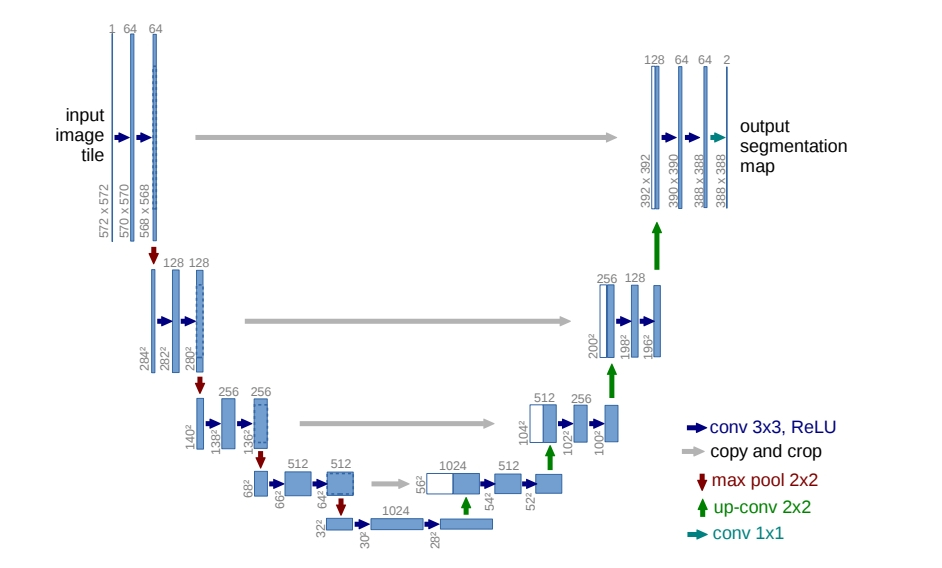
- Contraction Path는 우리가 아는 전형적인 CNN으로 구성되어 있다.
- 다만 위치 정보를 유지하기 위해 fully connected layer는 사용하지 않는다.
- 두 번의 3x3 convolution(unpadded) 수행 후 ReLU 활성화 함수를 적용한다.
- 한 번의 2x2 max-pooling(stirde 2)을 거친다.
- 정보 손실을 최소화하며 복잡한 특징을 잘 학습하기 위해 downsampling을 할 때 마다 채널의 수는 2배씩 증가한다.
- 요약하자면 이미지의 사이즈가 줄어드는 과정에서 이미지의 특징이 추출되고,
- 채널이 늘어나는 과정에서 이미지의 다양한 특징을 확보한다.
2. Expansive Path

- Expansive path에서는 2x2 up-convolution을 통해 feature map의 사이즈를 키우는 upsampling을 수행한다.
- 이 과정에서 채널의 수는 다시 절반으로 점차 줄어든다.
- 반대쪽 contracting path에서 같은 층에 있는 feature map을 가져와 부착한다.
- 이때 테두리는 cropping하여 가져온다.
- 두 번의 3x3 convolution(unpadded) 수행 후 ReLU 활성화 함수를 적용한다.
- 마지막 단에는 1x1 convolution을 사용해 결과값의 필터 수를 원하는 값(2개)으로 조정한다.
- 각 channel은 각각 세포, 세포막 classification에 사용된다.
Expansive path에서 사용되는 up-convolution에 관한 내용은 다음의 링크에서 살펴볼 수 있다.
[참고] CovTranspose2d에 관한 이해 자료 (upsampling) : https://westlife0615.tistory.com/257
[ pytorch ] ConvTranspose2d 알아보기
- 목차 키워드.- Convolution- CNN- AutoEncoder 들어가며.CNN 이나 AutoEncoder, VAE (Variational Auto Encoder) 등을 구현할 때에 Convolution Layer 을 위해서 Con2d 레이어가 사용됩니다.Convolution Layer 로 구성된 Neural Netwo
westlife0615.tistory.com
3. 입력 이미지에 적용한 기법들
Overlap tile & Mirroring the input image
U-Net 구조를 살펴보면, input image 사이즈가 572x572인 반면 output image 사이즈는 388x388이다. convolution layer에서 padding을 사용하지 않아 feature map 크기가 입력 데이터 크기보다 작아지며 입력 이미지의 가장자리 정보 점차 소실된다.
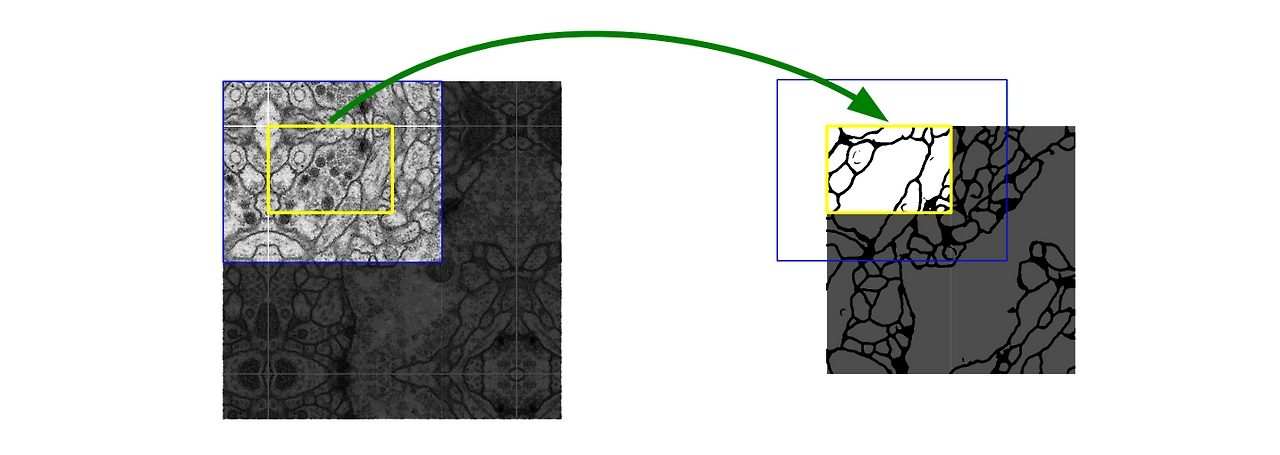
이에 U-Net에서는 "Mirroring extrapolation" 기법을 함께 사용하여 입력값으로 사용한다. Mirroring extrapolation은 입력 이미지의 가장자리를 "거울반사"된 형태로 확장하는 것이다. 이를 통해 missing data를 최대한 줄이고자 했다.

또한, 이미지를 tile로 나눠서 입력을 할 때 이전 입력의 일부분이 포함되어야 하기에 이를 overlap tile 기법이라고 말한다. 이러한 기법들을 통해 U-Net은 이미지 경계에서 패딩을 사용했을때 발생할 수 있는 정보 손실을 최소화할 수 있다.
4. Data Augmentation
본 논문에서는 Shift, Rotation, Gray value, Elastic Deformation 기법 등을 통해 데이터셋을 증강했다. 본 논문에서는 random-elastic deformation이 작은 데이터 셋을 이용한 학습에서의 key concept이라고 말한다. 여기서 random-elastic deformation은 pixel이 랜덤하게 다른 방향으로 뒤틀리도록 변형하는 방식을 말한다.

위의 그림을 보면 elastic deformation을 거쳤을 때 기본 이미지의 특성은 어느정도 유지되면서 이미지가 다양하게 변형되는 것을 볼 수 있다.
5. Training & Experiments
Training
- 인풋 이미지와 그 이미지의 segmentation map 이미지가 네트워크 학습 데이터로 사용됨
- SGD(Stochastic Gradient Descent, 확률적 경사 하강법) 사용하여 모델 최적화
- GPU Memory 사용 극대화를 위해 이미지를 타일로 분할(Tiling)하여 mini-batch로 구성한 뒤 모델에 입력
- Momentum 0.99 설정하여 이전 Training Sample에서 본 것들이 현재 Optimization Step에 영항을 많이 주도록 함
[Training Detail]
convolution을 계산할 때 패딩 없이 진행이 되기 때문에 이미지가 너무 작은 경우 경계값 정보가 많이 손실되어 학습이 제대로 이루어지지 않을 위험성을 가지 있기 때문에 여러 개의 작은 타일로 나누는 것보다 타일 크기를 크게 해서 전체 타일 수가 적은 것을 선호한다고 한다.
그러나 큰 타일 크기를 사용하면 GPU 메모리가 한정되어 있으므로 그만큼 SGD(Stochastic Gradient Descent) 배치 수는 줄어야 한다. 배치 수가 줄어든다면 그만큼 학습할 수 있는 데이터 다양성도 함께 줄어드는 단점이 있습니다. 즉 경사 하강법 과정에서 그래디언트 변동성이 커질 수 있게 되고, 이는 최적화 과정을 불안정하게 만들 수 있다.
따라서 논문에서는 이러한 문제점을 해결하기 위해 높은 모멘텀 momentum(0.99)을 사용하고 있다. 높은 모멘텀을 사용하면 변동성을 완화하고, 모델이 더 안정적으로 최적화를 할 수 있다.
모멘텀은 Local Minimum에 빠지는 경우를 대처할 수 있으며, 높은 모멘텀은 현재 그래디언트 업데이트에 이전 단계들의 그래디언트 영향을 더 많이 반영한다.
Loss function
Training에 사용한 U-Net의 Loss Function은 각 픽셀에 대한 에너지 함수(E)의 총합으로 구성되며, 에너지 함수는 다음과 같은 과정으로 계산된다.
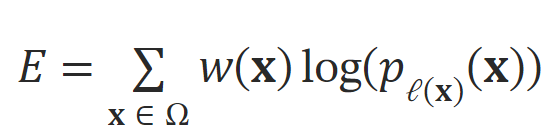
- w(x) : 각 픽셀마다의 weight
- l(x) : 픽셀 x의 실제 라벨
- :모델이 픽셀 x가 실제 라벨 l(x)에 속한다고 예측한 확률(Softmax 함수를 통해 계산)
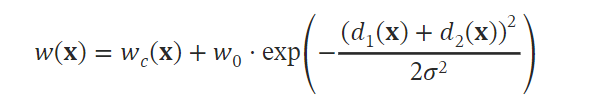
- w(x) : 각 픽셀마다의 weight
- w_c(x) : 클래스 빈도의 불균형을 해결하기 위해 도입(드물게 나타나는 클래스의 픽셀에 대해 더 높은 가중치)
- d1(x) : 픽셀 x의 위치로부터 가장 가까운 경계와 거리
- d2(x) : 픽셀 x의 위치로부터 두 번째로 가까운 경계와 거리
- 세포 경계 부분에서의 분류 오류에 대해 모델이 더 큰 손실 값 부과함으로써, 세포 경계를 더 정확하게 분류
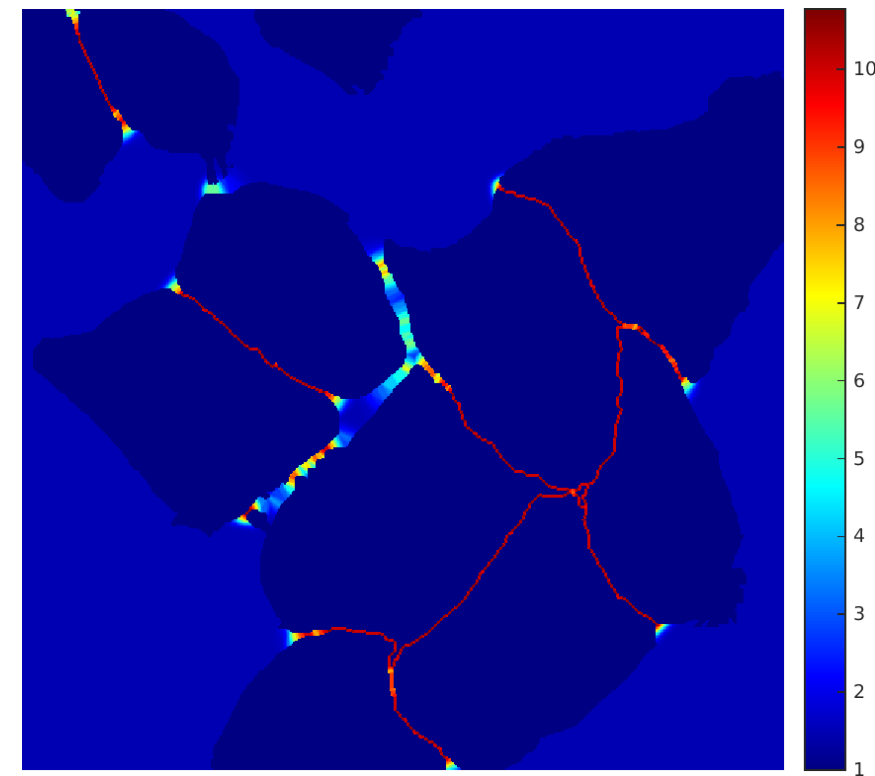
세포 분할 작업에서는 같은 클래스의 서로 접촉하는 객체를 분리해야 한다. U-Net은 이러한 문제점을 해결하고자 접촉하는 세포 사이의 배경 레이블에 큰 가중치를 부여하는 weighted loss를 사용한다. 탐지된 객체가 서로 가까이 붙어 있을 수록 그 사이의 배경이 큰 weight값을 갖게 된다. 위의 Fig3을 보면 세포 사이의 거리가 가까울수록 배경이 빨간 값을 가지게 된다.
[Loss Function Detail]
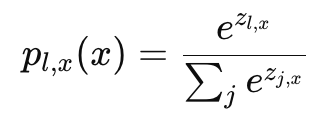
𝑙은 특정 클래스의 레이블을 의미

특정 클래스 𝑙에 대한 로그 확률

loss function
- 각 픽셀의 예측값에 SoftMax 함수를 적용해 예측값을 확률값으로 변환해 준다.
- 그런 다음, 특정 클래스 𝑙에 대한 로그 확률을 계산한다. 예측 확률은 0과 1 사이의 값을 가지게 되므로, 예측 확률이 1에 가까워질수록 로그 값은 0에 가까워지고, 예측 확률이 0에 가까울수록 로그 값은 매우 큰 음수가 된다.
- 각 픽셀의 실제 클래스 레이블과 예측된 확률 값을 비교하여 손실 값을 계산한다. 이때 Cross-Entropy Loss을 사용한다. 크로스 엔트로피에 픽셀 고유의 weight 𝑤(𝑥)을 곱함으로써 픽셀의 Loss값을 계산한다.
- 𝑤(𝑥) 식은 경계선(border)라인에 더 강한 학습을 시키기 위해서 가우시안 분포(Gaussian distribution)을 가정하고 경계선 근처의 픽셀에 더 높은 가중치를 부여하여 학습이 집중되도록 한다.
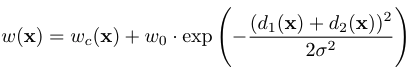
모든 픽의 에너지 함수(E)를 합산하여 전체 이미지에 대한 손실을 계산한다.
Experiments
[EM segmentation challenge]
- 전자현미경으로 찍은 세포 구조를 segmentation하는 대회
- Wrapping Error : 객체의 경계가 잘못 감싸졌거나 포장되었을 때 발생하는 오류
- Rand Error : 임의의 두 픽셀이 같은 객체에 속해야 하는지 혹은 다른 객체에 속해야 하는지를 정확하게 예측 못한 비율
- Pixel Error: 모델이 픽셀의 클래스를 잘못 분류한 비율
- U-Net이 Rank 1위

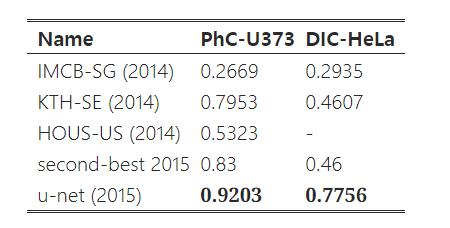
[ISBI cell tracking challenge]
- 광학 현미경에서 얻은 이미지로도 segmentation 수행
- 성능을 IOU로 측정
- IOU : 세포라고 구분한 영역과 실제 세포의 영역이 겹치는 정도
- 92%와 77%로 가장 좋은 성능
6. Conclusion
- U-Net은 다양한 biomedical segmentation applications에서 매우 좋은 성능을 보여준다.
- elastic deformation이 포함된 Data augmentation 덕분에 적은 수 데이터셋만으로도 훈련 가능
- 또한, NVidia Titan GPU(6GB)에서 단 10시간 만에 훈련시켜 합리적인 학습 시간을 가짐
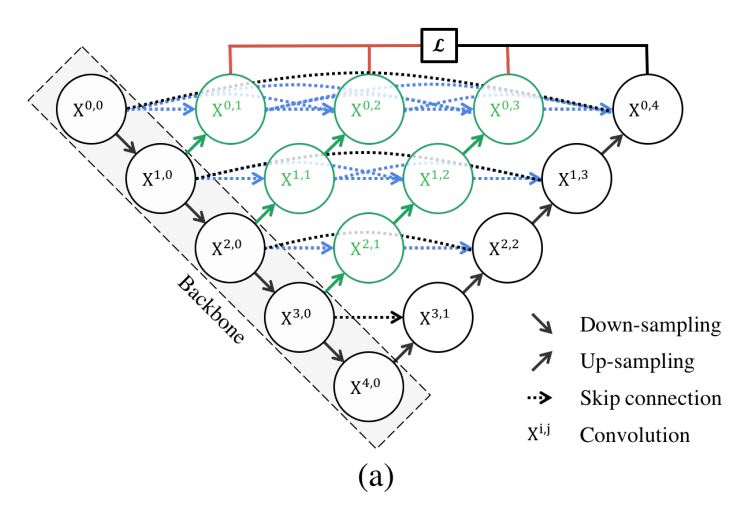
7. U-Net++
U-Net에서는 같은 해상도의 feature를 skip connection으로 한 번에 연결하여 encoder feature와 decoder feature 사이에 semantic gap(의미 수준 차이)이 크다. 이에 U-Net + “중간 단계들을 촘촘히 채운 skip connection”을 한 것이 U-Net++이다.
참고
https://hyeon827.tistory.com/37


