https://arxiv.org/pdf/2409.11169v2
https://build.nvidia.com/nvidia/maisi
maisi Model by NVIDIA | NVIDIA NIM
MAISI is a pre-trained volumetric (3D) CT Latent Diffusion Generative Model.
build.nvidia.com
Abstract
- MAISI (Medical AI for Synthetic Imaging) : 3D 컴퓨터 단층촬영 (CT) 이미지 생성 모델
- Volume Compression Network : 고해상도 CT 이미지 생성
- Latent diffusion model : flexible volume dimensions과 voxel spacing 제공
- ControlNet : Organ segmentation, Annotated synthetic image 생성
- 영역별, 조건별로 해부학적 segmentation image 생성, 생성 이미지를 통한 다양한 과제 해결
1. Introduction
Medical analysis image ML model 개발의 한계점
- 데이터 희소성 : 희귀 질환에서 제한된 데이터로 학습 미흡
- Human annotaion 비용 : 미묘한 진단에 필요한 전문 지식
- Privacy 문제 : 환자 정보에 민감한 윤리적 문제
Generaing synthetic data : 의료 이미지를 인공적으로 생성
- Data augmentation, 환자 데이터 의존성 ↓, cost-effective annotation alternative
- Generative model의 최근 발전을 Medical image에 적용 : multi-contrast MR/CT image synthesis, cross-modality image translation, image reconstruction
이전 연구의 한계점
- 고해상도 3D volume 생성의 어려움 : 3D framework의 엄청난 메모리 소비, Memory bottleneck 극복 필요
- 고정된 output volume dimensions, voxel spacing : 서로 다른 작업에서의 다양한 requirements와 비호환, 수치 조정에 대한 flexibility, utility 필요
- Data와 target organ에 대한 specialized model : 확장된 application에서 일반화 불가, 다양한 조건에 적용하였을 때 retraining의 필요성이 완화된 모델 필요
→ 고해상도 3D CT volume generation을 위해 Volume Compression Network, Latent Diffusion model, ControlNet으로 구성된 3D network 제안
- Volume compression network : 대량의 3D 의료 데이터를 latent space로 압축, Visual encoder, Visual decoder를 통해 latent feature를 mapping, Memory footprint를 줄이기 위한 시도로 TSP (tensor splitting parallelism) 도입
- Latent diffusion model : Latent feature 생성 용이, Flexible dimension 및 Condition(Body region, voxel spacing) 설정으로 낮은 메모리 소비를 유지하면서 복잡한 해부학적 구조 생성, Generalizability와 robstness를 확보하여 다양한 임상 시나리오 학습
- ControlNet : Output에 대한 제어, 광범위한 작업에 대한 versatility, applicability 향상, Retraining의 필요성 줄임, 시간 계산 리소스 보존

2. Related Work
- 이전에는 example-based approach, geometry-regularized dictionary learning 등 기존 영상 처리 기술 기반, but realistic, diverse한 생성 능력 제한
- Machine learning, Deep learning 출현으로 정교하고 정확한 모델 가능
GAN
- MRI / CT 이미지 합성과 같은 다양한 작업에 널리 채택 (cross-modality image translation, image reconstruction, super-resolution)
- Data augmentation by annotated image : complexity와 3D 특성 무시로 인해 2D medical image와 small volumetric patch synthesis에 한정
DM
- 고품질 영상 합성, 안정적 학습과정, conditioning의 유연성
- 복잡한 detain 포착과 최소한의 artifacts로 임상 사용에 적합한 고품질 2D medical image 생성
- GenerateCT : medical text prompts에서 3D CT volume 합성, slice의 순차적 생성, but slice 간 3D 구조적 불일치 문제
- DiffTumor : Various organ tumor segmentaion model의 robusness와 generalizabilty 향상
3. Methodology
- Volume Compression Network(VAE-GAN) : 고해상도 3D medical image를 latent space에 효과적으로 압축, memory usage와 computation complexity 줄임
- Latent diffusion model : Compressed latent space에서, body region, voxel spacing을 조건으로, flexible dimension에서 3D 해부학적 구조에 대한 feature 생성
- ControlNet : trained latent DM의 2nd stage에서 추가 condition 주입하여 output을 dynamic control, 광범위한 작업에 적용 가능, 서로 다른 task에 적용 시에도 retrianing 필요 감소
3.1 Volume Compression Network
- VAE(Variational Autoencoder) 적용 : combined objectives로 학습 (perceptual loss $\mathcal{L}_{lpips}$, adversarial loss $\mathcal{L}_{adv}$, L1 reconstruction loss $\mathcal{L}_{recon}$)
- Volume reconstruction이 image manifold에 밀접하게 부합하고 local realism 강화
- KL regularization $\mathcal{L}_{reg}$ : high-variance latent space 피함
$x\in\mathbb{R}^{H\times W\times D}$
$z=\mathcal{E}(x) \in \mathbb{R}^{h \times w \times d}$
$\tilde{x}=\mathcal{D}(z)=\mathcal{D}(\mathcal{E}(x))$
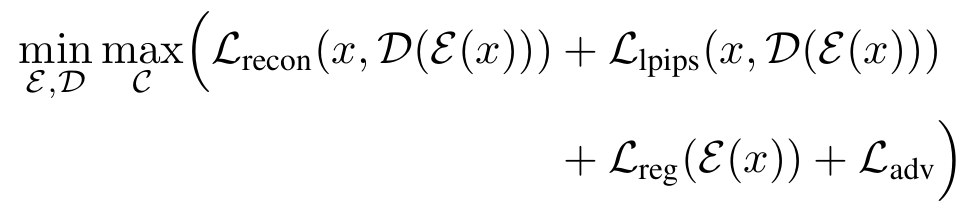
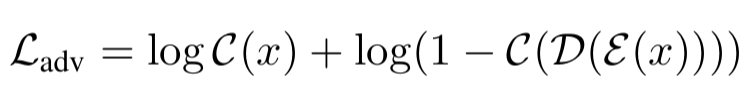
Tensor spliting parallelism (TSP) : Memory bottleneck 해결
- 2D 고해상도 이미지 생성에서 사용한 additional super-resolution model은 부족 (GPU memory limitation)
- Sliding window inference : smaller 3D patch 분할 후 각 output을 stitching, window boundary에서 artifact/discontiuities 문제가 두드러짐
- Convolution, normalization layer에서 필요한 overlap을 보존하면서 feature map을 더 작은 segment로 분할, 지정된 device에 할당 후 병합되어 layer output 생성
- Inference 가속화, 각 segment를 하나의 device 내에서 순차적으로 처리하여 최대 메모리 사용량 줄임

3.2 Diffusion Model
- flexible dimension의 압축된 latent space에서 작동, body region 및 voxel spacing을 conditional input으로 통합
- Data distribution $p(x)$에서 denoising 작업을 거쳐 생성되는 과정을 학습, Markov chain
- $\epsilon_{\theta}$는 denoising learning model로 time-conditional U-Net, $z_t$는 input latent feature의 denoised version
- Body region : $i_{top}$, $i_{bottom}$ (촬영할 영역을 나타내는 one-hot vector)으로 작업할 CT 영역을 나타냄
- Voxel spacing : $s$ (voxel의 크기 3차원 vector)
- $c_p=\left\{ i_{top}, \, i_{bottom}, \, s\right\}$
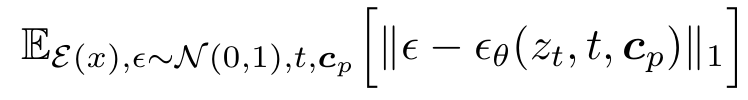
- $\epsilon_{\theta}$는 전 training process에서 dimension이 달라지는 latent variable $z_t$ 학습, flexible volumetric dimension의 output 생성 가능
3.3 Additional Conditioning Mechanisms
- 생성된 output의 제어 및 flexibility 향상 : auxiliary condition을 diffusion process에 주입하여 precise control 구현
- Locked copy (original model knowledge 보존), trainable copy (특정 조건에 맞게 학습 가능), zero convolution layer
- Compact encoder network를 사용하여 입력된 condition을 latent feature 내 task-specific condition $c_f$로 전환
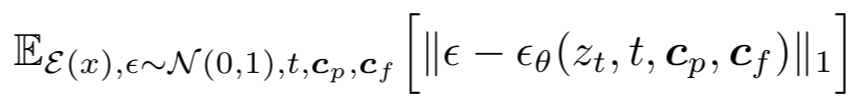
- specific needs of various medical imaging task를 retraining 과정 없이 fine-tuned된 모델 생성
4. Experiments
4.1 Datasets and Implementation Details
3개의 구성 모델(VAE, DM, ControlNet)별로 적절한 데이터셋에 대해 학습. 정상 범위 내 주요 장기가 잘 생성되었는지 품질 확인
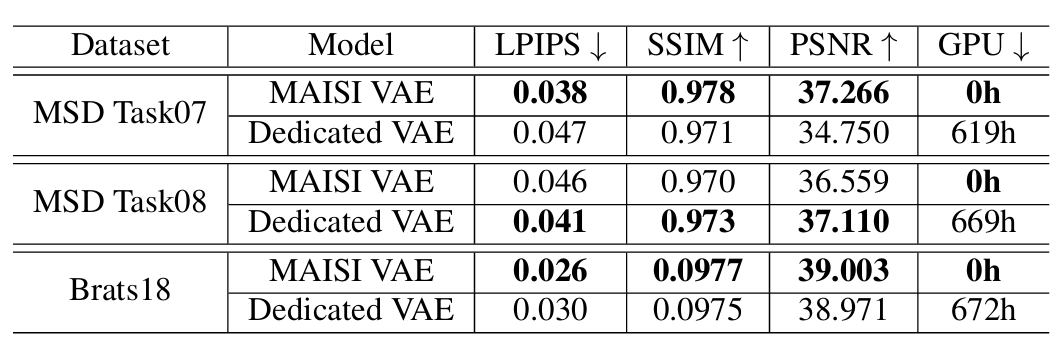
4.2 Evaluation of MAISI VAE
- out-of distribution dataset에 대해 MAISI VAE model과 dedicated VAE(test dataset 내에서 학습) 성능 비교
- 추가 GPU resouce 지출 없이 비슷한 결과 달성, 모델 효율성과 실용성 및 최적화 잠재력을 보여줌.
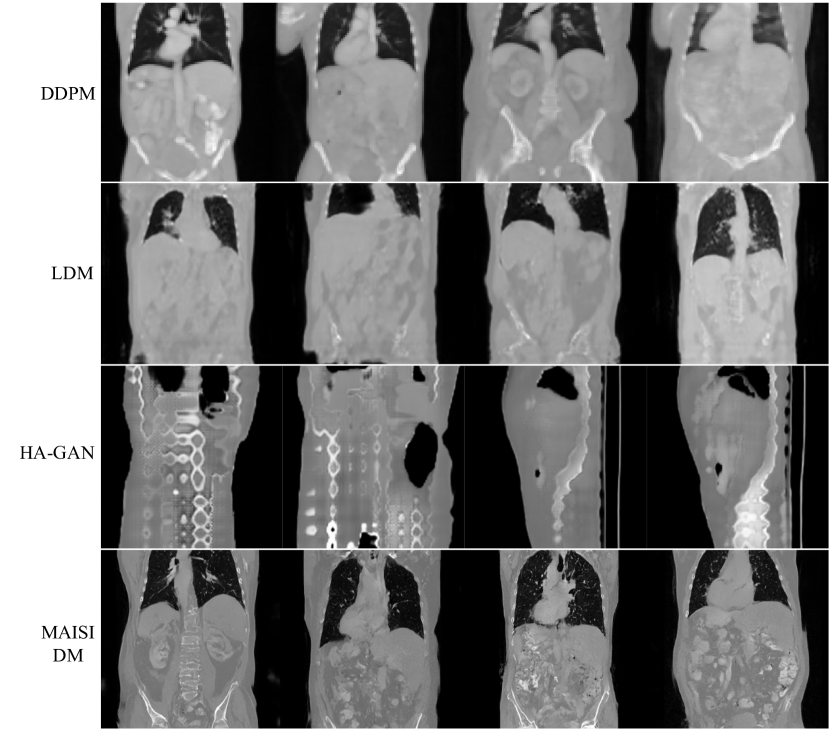
4.3 Evaluation of MAISI Diffusion Model
- Synthesis quality : 실제 dataset과 비교하였을 때 FID 수치 비교
- DDPM, LDM, HA-GAN과 비교하였을 때 실제 dataset과 유사한 이미지를 생성.


Response to primary conditions : Body regions, voxel spacing condition에 맞게 높은 flexibility와 control을 보여줌.

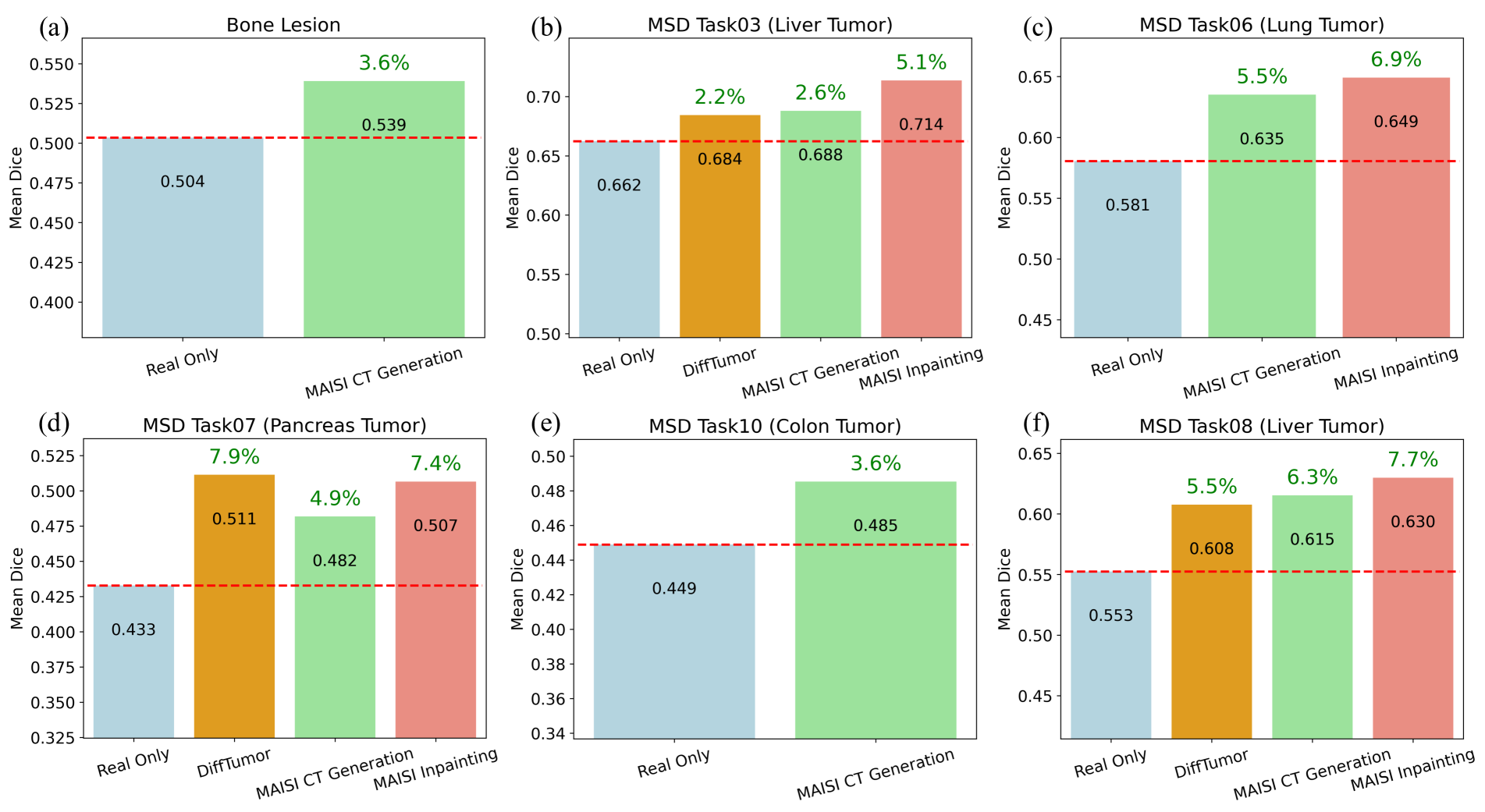
4.4 Data Augmentation in Downstream Tasks
- Real data로만 구성된 dataset(Real Only)과, Real data에 synthetic data by from model을 섞은 dataset으로 훈련된 segmentation model의 DCS 수치 비교
- MAISI CT Generation (단순 생성), MAISI Inpainting(건강한 환자의 real data에 tumor 합성)
- MAISI dataset augmentaion에서 DSC 향상, out-of-distribution dataset에 대해 test를 수행했을 때 높은 성능을 보임.
5. Discussion and Limitation
- 고품질 CT 이미지를 생성하는 데 큰 잠재력
- 한계 : 인구 통계적 변화 표현, 여전히 상당한 계산 리소스
6. Conclusion
- Foundation model (VAE + LDM)과 ControlNet의 조합으로 고해상도 3D CT Volume을 생성하는 모델
- 해부학적으로 정확한 이미지 생성에 대한 adaptable, versatile한 solution
- Flexible volume dimensions와 voxel spacing으로 사실적인 CT 이미지를 생성하고, Medical dataset에 대한 data aumentation을 수행하여 downstream task의 preformance 상승에 기여




