https://arxiv.org/abs/2403.00771
XProspeCT: CT Volume Generation from Paired X-Rays
Computed tomography (CT) is a beneficial imaging tool for diagnostic purposes. CT scans provide detailed information concerning the internal anatomic structures of a patient, but present higher radiation dose and costs compared to X-ray imaging. In this pa
arxiv.org
Abstract
- Computed tomography (CT) : 해부학적 구조를 반영한 detailed information. Radiation dose, Cost ↑
- Orthogonal X-ray를 CT volume으로 변환하는 이전 연구 → 더 큰 dataset과 다양한 model structure
- UNet architecture, custom connection, activation function, loss function, optimizers, back projection 접근
1. Introduction
Computed tomography (CT)는 환자 신체를 다양한 각도에서 찍은 X-ray 이미지를 재구성한 것으로, detail한 해부학적 구조를 취득할 수 있다. 이러한 정보들은 진단 과정에 도움이 되지만, resource-demanding, high-levels of ionizing radiation exposure의 문제를 지닌다. 반대로, X-ray는 resource-efficient, lower average radiation dose라는 효율적인 특성으로 널리 활용되는 image modality이지만, detailed 3D spatial information이 부족하다는 단점을 지닌다.
많은 Healthcare protocol에서 X-ray는 preliminary information을 모으기 위해 사용되지만, bone structure와 달리 soft tissues 인지에는 취약한 모습을 보인다. Machine learning method는 2D X-ray를 3D로 변환시킴으로써, X-ray의 결점을 보완하여 well-informed decision을 할 수 있도록 추가 정보를 제공하고자 하였다.
논문의 저자는 X-ray image input으로부터 simulated CT scan을 생성하는 이전 연구들을 개선하고자 하였다. 최근 연구에서는 X2CT-GAN이라는 모델을 통해 orthogonal X-ray views로부터 simulation of CT scans을 생성하였는데, 이는 특수 generator를 사용하는 GAN framework를 이용하였다. 본 논문에서는 더 큰 CT dataset과 다양한 network architecture를 통해 생성되는 이미지의 품질을 개선하고자 한다. Chest region의 Paired X-rays를 CT로 재구성하는 deep learning model을 개발하였고, 이는 preliminary detection of abnormality 및 추가적인 resource-intensive imaging methods 사용 여부 결정에 보탬이 되도록 한다.
2. Data
- Rad-ChestCT dataset, CheXpert dataset 사용 (각각 분리된 CT, X-ray dataset)
- CT scale normalization, Volume resizing, Training vs validation set, test images 등 기법에 대해 설명
3. Models
Simulated X-ray image가 reconstruction model input으로 필요
→ Style Transfer model : Experiment 수행을 위해 각 Dataset에서 paired X-ray data 생성
3.1 Style Transfer General Adversarial Network
- CheXpert와 Rad-ChestCT dataset에서 pair를 맞춰야 함.
- CycleGAN : Unpaired dataset mapping을 학습 가능한 model
- Style transformation model : Real X-ray image style을 학습하고 mean CT scan 형태로 바꿀 수 있도록 함. → Paired data generation
- Mean CT scan : coronal, sagittal axis 평균을 내어 2D CT scan 생성, style transfer GAN의 input이 되어 X-ray image style과 비슷하도록 변환됨 → Simulated X-ray와 CT scan의 pair 생성
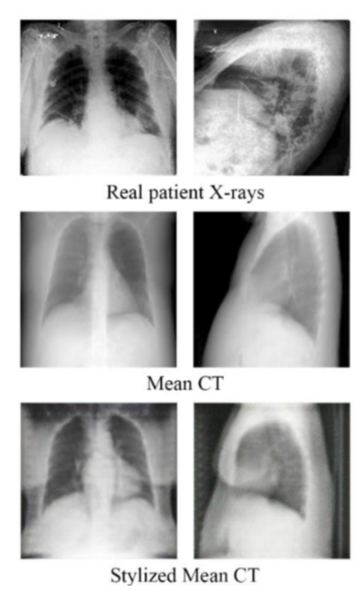
- CycleGAN style transformation이 더욱 효과적으로 이루어지도록 model 변경
- Learning rate ↓ : X-ray to CT는 non-linear한 contrast 변환 작업, EMA(exponential moving average)를 적용하여 가중치를 해당 평균으로 재설정 → fine level update, generalization 성능을 높임
- Nadam optimizer : Adam 대신 적용, local minimum에서 overshooting 문제 줄임 → 더 나은 convergence 성능
- $\lambda=20$으로 증가 : cycle consistency loss 조절, original input과 얼마나 유사한지 평가하는 지표
(기본값은 10, $\lambda$↑ pixel alignment 개선, $\lambda>25$에서 입출력간 차이가 거의 없어짐 (transformation 성능 저하))
→ 주요 X-ray 특징을 유지하면서 원본 mean CT와 유사한 형태를 유지
3.2 Reconstruction Model
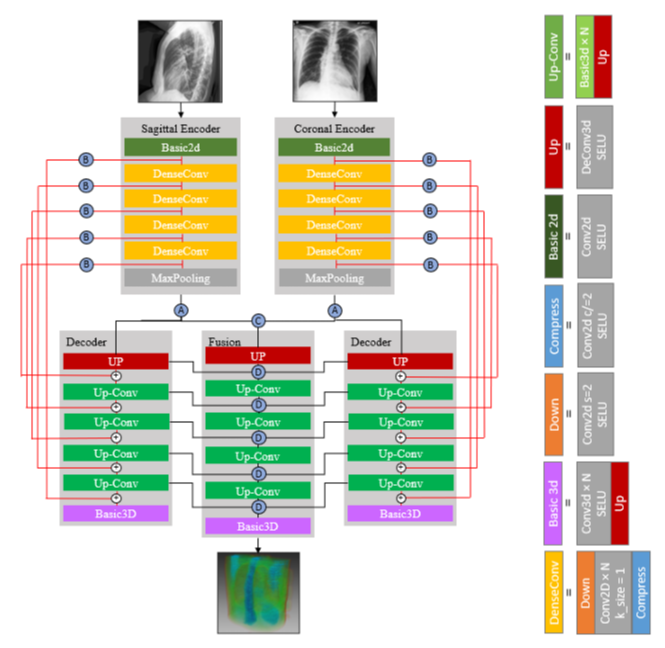
- 2개의 orthogonal X-ray를 simulated CT 3D volume으로 변환
- X2CT-GAN 논문에서 제안된 X2CT-CNN+B 모델에서 영감
- 2개의 UNet encoder-decoder architecture, 각각 simulated X-ray 입력을 처리
- Fusion network 존재 : Decoder 단계에서 skip connection을 적용, convolved feature를 반복적으로 추출하여 최종적으로 CT volume을 생성
- Modification : Experiment 과정에서 architectural, hyperparameter 변화
- 이전 연구와 다르게, 일부 experiment에서 real X-ray - CT scan 사용하기도
- Keras Tensors bit limitation : 256×256 pixel에서 128×128 pixel image 사용, 128×128×128 voxel CT scan 사용으로 memory 절약
- UNet : skip connection을 통한 input image feature 보존 능력 우수, medical-imaging application에서 작은 detail 보존이 매우 중요
3.3 Reconstruction Model Connection

- Reconstruction model component를 연결하는 Connection. A~D 4가지로 구성.
- Experiment 과정에서 connection의 위치와 I/O는 변하지 않았지만, connection의 내부 정의만 수정. 전체적인 구조를 유지하면서 세부적인 모델 변화를 테스트하기 위해 수행
- Connection-A : input tensor를 3D output으로 변환
5차원 Tensor (batch size, rows, columns, depth, channels) - Connection-B : UNet의 Skip connection
Conv2D와 Conv3D layer를 통해 channel 수를 맞춰주고 2D encoder image를 3D decoder volume으로 변환 - Connection-C : sagittal - coronal voxel 표현 변환. Fusion network decoder input
sagittal view가 front-facing coronal view와 직교하도록 조정 후 평균화하여 voxel 표현 - Connetion-D : Connection-C의 구조를 통해 도출된 average output과, fusion decoding layer에서 추가 input을 받아 concatenated
4. Experiments & Results


4.1 Input X-ray Dimensions
- Various input dimensions : output도 비슷한 크기로 matching
- 128×128 : X2CT-CNN+B와 동일
- 64×64 : memory, training time을 줄였으나, artifacts가 존재하며 key feature, pattern 인지에 실패
4.2 Training Loss Function & Optimizer
- 초기 Experiment의 loss function과 optimizer 선정은 이전 연구 guideline을 참조하여 MSE, Adam으로 수행
→ Noisy output : Deep learning model deficiency in adequate capacity - Key feature 간 variation은 부적절한 loss function 사용에서 기인. MSE는 spatial awareness가 부족하다고 판단함.
- MAE가 blurriness를 줄이는데 최적 : Output feature precision은 줄어들지만, preliminary imaging에는 적합
- Learning rate optimization은 Adam으로 시작했으나 local minima escaping에 어려움
- Nadam을 통해 Convergence issue 최소화 : Generalization에 적합, 가장 clear한 image output 도출
- MAE, Nadam은 blurriness를 줄이기 위해 선정, 기존 방법의 similarly를 수행하는 데도 문제없음
- MAE : linearly loss prediction, 각 error값을 동일한 importance로 적용
- Nadam : Nesterov acceleration - momentum weight을 future location에 맞춰 optimization 수행
4.3 Connection-A Architecture
- 64-Dense vs 512-Dense : Connection A architecture를 달리하여 비교, Apex Unet의 dense-layer neuron 개수 변화
- Visual, quantitative difference는 차이가 없으나, 512-Dense가 outside the intrathoracic cavity feature에 더 aware (shoulder blades, upper lower vertebrae)
4.4 Back Projection Injection
- Orthogonal X-ray images로부터 back projection : spatial data를 보존할 수 있는 계산 결과이며, 전통적 CT reconstruction method로 이용됨.
- Injection 수행 시 Granular, key features 향상
5. Challenges
5.1 Memory Limitations of Single-GPU Training
- Layer 개수와 image 크기가 run-time 동안 많은 메모리를 소비 (final output size가 가장 큰 원인)
- High pixel input, output으로 accuracy를 향상시키려 하지만 memory limit 때문에 최대 품질이 제한됨
- Reconsturuction model은 25GiB, Projection injection은 32GiB 소비
5.2 TensorFlow's Max Limit on Parameters
- Tensorflow의 layer당 max parameter limitation ($2^32$ parameters per layer)
- 가능한 max ouput size에 영향, 최대 품질이 제한됨
- Keras의 layer limitation 준수하여 experiement 수행, PyTorch를 활용하여 해결 기대
6. Future Work
6.1 Switch to PyTorch to Solve Max Parameters Constraint
TensorFlow의 Keras layer parameter limitation : 더 많은 parameter 필요. PyTorch library로 변경하여 코드 조정 필요
6.2 Transformer Architecture
X-ray image를 직접 encoder에 입력하는 구조, multi-headed attention layer 추가 시 품질 향상 기대
6.3 Multi-GPU Training
단일 GPU로 training 시 학습 속도 저하로 충분한 epoch동안 학습되는 것을 제한
Multiple GPU 환경에서 학습 시간 단축 가능, 더 많은 epoch 동안 model 훈련하여 성능 개선
6.4 Use Medical Label AI to Test Accuracy of Output
Rad-ChestCT와 CheXpert dataset에서 동일한 label을 가진 data끼리 matching
정보 소실없이 CT scan을 적절하게 재구성
6.5 Train on Larger-scale X-Ray Images
Input X-ray 크기 제한 → CT maximum quality 저하
Input image를 256×256 이상으로 확장 필요
6.6 Partner with Medical Experts to Evaluation Usability
의료 전문가와 협력하여 모델을 평가하고, 임상 환경에서의 관련성을 검토
모델의 개선이 필요한 부분을 식별하고 향후 연구 방향을 제안
7. Conclusion
- Paired orthogonal X-rays로부터 CT scan을 재구성하는 다양한 technique에 대해 탐구 : Reconstruction model을 통해 simulated CT volume 생성
- Style transformer model로부터 만들어진 simulated X-ray를 사용한 experiment가 우수한 성능, Back projection model이 더 정확한 granular featrue 구현
- Multiple GPU, attention 추가, input X-ray image size 향상 등 추가 연구 필요
- Medical experts에게 feedback을 받고 CT labeling model을 통해 평가 필요




