[2025-2] 최민서 - 3D-HLDM: Human-Guided Latent Diffusion Model to Improve Microvascular Invasion Prediction in Hepatocellular Carcinoma
[논문링크] https://ieeexplore.ieee.org/document/10635195/authors#authors
3D-HLDM: Human-Guided Latent Diffusion Model to Improve Microvascular Invasion Prediction in Hepatocellular Carcinoma
Microvascular invasion (MVI) is a critical risk factor for survival in patients with Hepatocellular Carcinoma. The presurgical prediction of MVI is clinically important and crucial for surgical and treatment planning. Although deep learning models have bee
ieeexplore.ieee.org
1. Introduction
Microvascular Invasion(MVI)의 유무를 예측하는 것은 의학적 관점에서 굉장히 중요하다. 따라서 환자의 MRI 데이터를 이용하여 MVI 유무를 예측하는 방법이 연구되었다. 이는 굉장히 복잡한 작업으로 전문가들 사이에서도 예측 결과가 다른 경우가 많다. 현재 해당 태스크에서 가장 좋은 성능을 보이는 방법론은 딥러닝을 이용한 방법이다. 하지만 학습에 필요한 데이터의 개수가 적어 모델의 성능을 높이는 데에 제약이 되고 있다.
이러한 문제를 해결하기 위해 논문에서는 human-guided 3D Latent Diffusion Model(3D-HLDM)을 제안한다. 이는 Latent Diffusion Model(LDM)을 학습시켜 원본 데이터(3D MRI 사진들)과 유사한 이미지들을 생성하도록 한 후, 생성된 데이터들을 포함하여 데이터셋을 구성하여 MVI 유무 예측 모델을 학습시키는 방식이다. 추가적으로 LDM의 학습 과정에 강화학습의 방법론을 융합하여 생성되는 데이터의 품질을 높인다.
2. Methods
2.1 Data preparation
학습을 위한 데이터로 삼성 메디컬 센터에 있는 494명의 익명의 환자들의 MRI 스캔 결과를 사용했다. 각 데이터들은 수년간의 진찰 결과를 바탕으로 MVI 유무가 분류되었다. 구체적으로, 각 MRI 스캔 데이터 중 HBP에 해당하는 것만 채택한 뒤, 2D DICOM slices를 추출하였다. 이후에 이미지 품질이 낮은 환자 19명의 데이터를 제외하고 2D DICOM slices를 단일 3D NIfTI 이미지로 결합하였다. 결과적으로 475명의 환자들이 최종 데이터셋에 포함되었으며, 274명은 MVI-positive, 201명은 MVI-negative으로 분류되었다.
빠른 학습과 개인정보 보호를 위하여 각 이미지에서 (96,96,48) 크기의 Region of Interest(ROI)를 잘라내어 학습에 사용했다. ITK-SNAP 소프트웨어를 이용해 lesion core의 3D 좌표를 얻어 ROI를 얻었다. 이 후 데이터가 0에서 1 사이의 값을 갖도록 정규화를 해주었다.
2.2 3D Latent Diffusion Model
논문에서는 원본 LDM 모델을 바탕으로 학습에 사용할 3D LDM을 설계했다. NVIDIA MONAI 소프트웨어가 사용되었다. LDM의 구체적인 구조는 아래와 같다.
- 오토인코더
인코더의 각 레이어는 64, 128, 256 채널을 가지며, (96,96,48) 크기의 이미지는 인코더를 거쳐 (8,24,24,12) 크기의 잠재공간에 임베딩된다. 디코더는 이와 대칭적으로 각 레이어가 256, 128, 64 채널을 가진다.
- U-Net
3D U-Net은 각 스텝에서 128,256, 512 채널을 가진다. 잡음 스케줄은 DDPM을 사용했고 1000 스텝을 적용했다.
2.3 Proximal Policy Optimization
논문에서는 학습된 LDM을 파인튜닝하기 위해 Proximal Policy Optimization(PPO) 방식을 적용했다. 이는 아래의 목적함수를 최대화하는 강화학습의 한 방식이다.
$$ L^{CLIP}(\theta) = \hat{\mathbb{E}}_t \[ \min(r_t(\theta)\hat{A}_t), \mathrm{clip}(r_t(\theta), 1-\epsilon, 1+\epsilon)\hat{A}_t \] $$
$\theta$는 정책 파라미터, $\hat{\mathbb{E}}_t$는 타임스텝 동안 계산된 경험적 평균을 의미한다. $r_t(\theta)$는 업데이트 전후 정책에 대하여 특정 행동을 취할 확률의 비율이다. $\hat{A}_t$는 추정된 advantage 값으로 행동의 기대보상값과 상황의 기대보상값의 차이이다. $r_t(\theta)$를 clip함으로써 PPO는 정책의 업데이트가 과도하게 되는 것을 막는다.
PPO를 3D diffusion model에 적용하기 위해 random noise를 상태(state)로 보고, 생성된 3D 이미지를 행동(action)으로 본다. 값을 0에서 1 사이로 정규화하면 이를 확률로 볼 수 있다.
2.4 Reward model
PPO는 $\hat{A}_t$를 계산하기 위해 Reward Model(RM)이 필요하다. 논문에서는 RM을 따로 학습시킨 후 PPO에 적용한다. RM의 학습은 아래와 같이 진행된다.
1) 학습된 LDM을 이용해 이미지를 생성한 후, 임의로 두 개의 이미지를 뽑아 MRI 이미지 쌍을 구성한다.
2) 전문가가 두 이미지 중 더 진짜 MRI 이미지 같은 이미지를 선택한다.
3) 아래 손실함수에 따라 RM을 학습시킨다.
$$ L(\theta) = -\frac{1}{N}\sum^N \log ( \sigma( r_\theta(i_c) - r_\theta(i_n) ) ) $$
위 수식에서 $N$은 데이터셋의 사이즈이고 $i_c$와 $i_n$은 각각 선택된(chosen), 선택되지 않은(not chosen) 이미지를 의미한다. 학습 시 메모리 제한으로 인해, mini-batch gradient descent와 gradient accumulation을 사용한다. RM 구조는 4개의 3D CNN 레이어로 구성된다.
3. Experiments
학습에는 NVIDIA A100 80GB GPU와 MONAI 1.10, Python 3.8이 사용되었다.
3.1 Latent Diffusion Model Training
LDM의 학습은 순차적으로 진행되었다. 먼저 오토인코더를 2000 epoch 동안 학습시켰다. 학습에는 $10^{-7}$의 KL weight, learning weight $10^{-6}$의 Adam optimizer가 사용되었다. 학습은 A100 GPU로 3일 걸렸다.
이후 10000 epoch 동안 3D U-Net을 학습시켰다. 학습에는 158명의 MVI-negative 환자들이 사용되었다. learning rate $10^{-5}$의 AdamW optimizer가 사용되었다. 학습은 A100 GPU로 2일 걸렸다.
3.2 Human feedback
3.1의 방식을 따라 학습된 LDM을 이용하여 1000개의 3D MRI 이미지를 생성했다. 이 이미지들 중 2개의 이미지를 랜덤으로 뽑은 다음에 전문가가 더 진짜 같은 MRI 이미지를 뽑는 시행을 반복한다. 총 3536쌍의 이미지 페어에 대해 선택이 진행되었으며 이 데이터를 이용하여 RM을 학습시켰다. RM의 학습은 100 epoch 동안 진행되었고 learning rate $10^{-4}$의 AdamW optimizer를 사용하였다. 2.4에서 설명한 것처럼, 학습 데이터를 12개의 미니배치로 나눈 후 gradient accumulation을 적용했다. 학습은 A100 GPU로 1시간 걸렸다.
3.3 Latent Diffuson Model Fine-tuning
학습된 RM을 이용해서 U-Net을 파인튜닝한다. 파인튜닝에는 PPO 알고리즘이 사용된다. 각 스텝마다, 하나의 MRI 이미지가 생성되고, RM은 생성된 이미지의 보상을 계산한다. 하나의 이미지를 생성하는 과정 전체를 하나의 에피소드라고 하자. 논문에서는 30 에피소드를 진행했다. PPO 손실함수를 통해 diffusion U-Net을 20 epoch 동안 learning rate $10^{-4}$와 $\epsilon=0.1$의 Ada, optimizer를 사용했다. 학습은 A100 GPU로 4시간 걸렸다. 학습된 모델로 이후 실험에 사용될 300개의 MRI 이미지를 생성했다.
3.4 MVI Prediction
학습된 3D-HLDM 모델로 생성한 이미지를 MVI 예측 딥러닝 모델에 데이터로 추가했을 때, 성능이 얼마나 향상되었는지를 실험을 통해 관찰하였다. 먼저 400명의 환자를 8:2로 나누어 training, validation set을 구성했고, affine 변환과 random rotation transformation을 적용했다. 그 후 구성된 데이터를 이용해 다양한 MVI 유무 예측 딥러닝 모델을 학습시켰다. 다음으로, 기존의 데이터셋에 3D-HLDM 모델로 생성한 300개의 MVI-negative 이미지를 추가해서 똑같은 환경에서 학습시켰다.
또한 성능 향상이 단순히 MVI-negative 데이터를 추가함으로써 얻는 편향 때문인지 측정하기 위해 VQVAE 모델을 학습시켜 편향의 영향을 측정했다. 먼저 각각 32차원을 갖는 256개의 임베딩의 discrete embedding space를 이용하는 VQVAE를 학습시켰다. 학습은 LDM과 같은 MVI-negative 데이터셋에서 300 epoch 동안 진행되었다. 학습된 VQVAE 모델을 이용해 진짜 MVI-negiatve 이미지를 인코딩-디코딩 하여 강화된 MVI-negative 이미지를 생성했다. 이후 3D-HLDM으로 생성한 300개의 이미지를 새롭게 생성된 강화 이미지로 대체하여 학습을 진행하였다.
MVI 예측 모델로는 ResNet, DenseNet, EfficientNet을 사용했다. 각 모델별로 깊이를 다르게 하여 총 12개의 모델에 대하여 실험을 진행했다. 모든 모델의 학습에서 patience 5의 early stopping을 적용했고 learning rate $10^{-4}$의 Adam optimizer를 사용했다. 학습은 A100 GPU로 모델당 1시간 걸렸다.
4. Results
4.1 MVI prediction performance
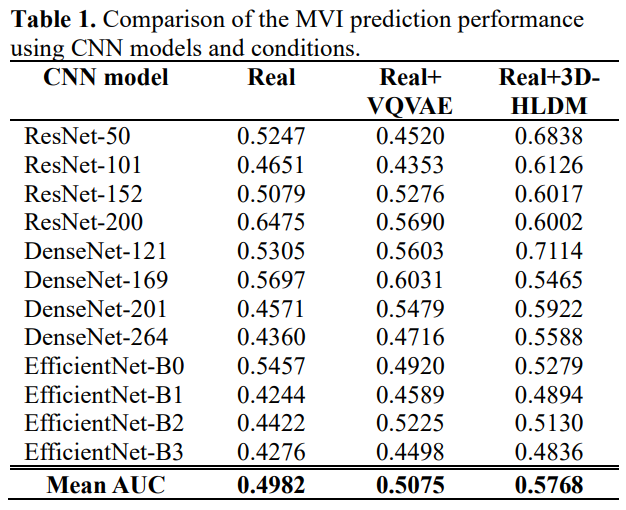
아래 표는 MVI 예측 실험의 결과를 보여준다. 대부분의 경우에 3D-HLDM으로 생성한 데이터를 학습한 모델이 더 좋은 성능을 보였다. AUC 점수의 평균도 다른 것들과 비교했을 때 확연히 높았다. 이는 Wilcoxon Signed-Rank Test를 통해 통계적으로 검증할 수 있었다.
4.2 High-resolution Synthetic MRI
아래 그림에서 볼 수 있듯이 학습된 3D-HLDM으로 생성한 MRI 이미지는 품질이 좋았다. 또한 3D-HLDM은 liver shape, lision size 등을 조절하여 다양한 이미지들을 생성할 수 있었다.

5. Discussion and Conclusion
논문에서는 의학 분야에서의 데이터 부족을 극복하는 새로운 방법을 제시했다. 3D LDM을 human feedback을 바탕으로
파인튜닝하여 높은 해상도의 MRI의 이미지를 얻어 MVI 예측 모델을 위한 데이터셋을 생성하는데에 사용하여 MVI 예측 모델의 성능을 유의미하게 높일 수 있었다.