[2025-1] 정유림 - Graph neural networks for single-cell omics data: a review of approaches and applications
Title: Graph neural networks for single-cell omics data: a review of approaches and applications
Published in: Briefings in Bioinformatics, 2025, Vol. 26(2), bbaf109
DOI: 10.1093/bib/bbaf109
논문링크 : https://academic.oup.com/bib/article/26/2/bbaf109/8080373
한 줄 요약 : GNN(Graph Neural Network)로 single cell omics data를 더 잘 분석할 수 있다.
Single Cell Omics Data란?
1. Single Cell (단일세포)
- 인체는 여러 조직으로 이루어져 있고, 그 조직은 수많은 세포로 구성되어 있음
- 기존에는 조직 단위(벌크)로 유전자 데이터를 분석했지만, 이제는 한 개의 세포 수준(single-cell resolution)으로 분석 가능
- 개별 세포마다 유전자 발현 상태, 염색질 구조, 단백질 양 등이 다름
- 이렇게 세포 하나하나에서 얻은 데이터를 single cell data라고 함
2. Omics (오믹스)
- 생물학적 정보를 전 범위적으로 포괄하는 분석 분야를 지칭하는 접미사
- 종류:
- scRNA-seq: 단일세포 유전자 발현 (transcriptomics)
- scATAC-seq: 염색질 접근성 (chromatin accessibility)
- scDNAm: DNA 메틸화 (epigenomics)
- scHi-C: 3D 염색체 구조 (chromosome conformation)
- spatial transcriptomics: 세포 위치 기반 유전자 발현
- proteomics: 단백질 발현
→ 이런 여러 층위의 생물학적 정보를 아우르기 때문에 "omics"라 부름
왜 중요한가? (Single-cell omics의 장점)
- 세포 간 이질성(heterogeneity) 분석 가능
→ 암세포와 정상세포가 어떻게 다르게 행동하는지 파악 - 희귀 세포 집단 탐지
→ 질병 초기에 존재하는 소수 세포를 포착 - 세포–세포 간 상호작용 추론
→ 면역세포가 종양세포와 어떻게 커뮤니케이션하는지 예측 가능 - 유전자–유전자 간 조절 관계 분석
→ 유전자 네트워크 구조 해석 (e.g., 전사 조절)
Single-cell omics 데이터의 단점
| Noise | 측정 오류 또는 생물학적 변동성에 의해 신호가 불안정함 |
| Sparsity | 많은 유전자에서 발현값이 0 (dropout 현상) |
| 고차원성 | 수천~수만 개의 feature가 존재해 해석이 어렵고 계산량이 큼 |
Graph를 이용하는 이유
이런 생물학적인 context에서는
- 세포와 세포의 상호작용, 유사성
- 유전자와 유전자 사이의 상호작용, 유사성
이 중요하다.
이때 이런 관계는 단순한 Euclidean space에 존재하는게 아니기때문에 복잡한 관계로 존재하는데,
GNN은 non-Euclidean structure를 다룰 수 있기때문에 적합하다.
Graph 데이터의 특징
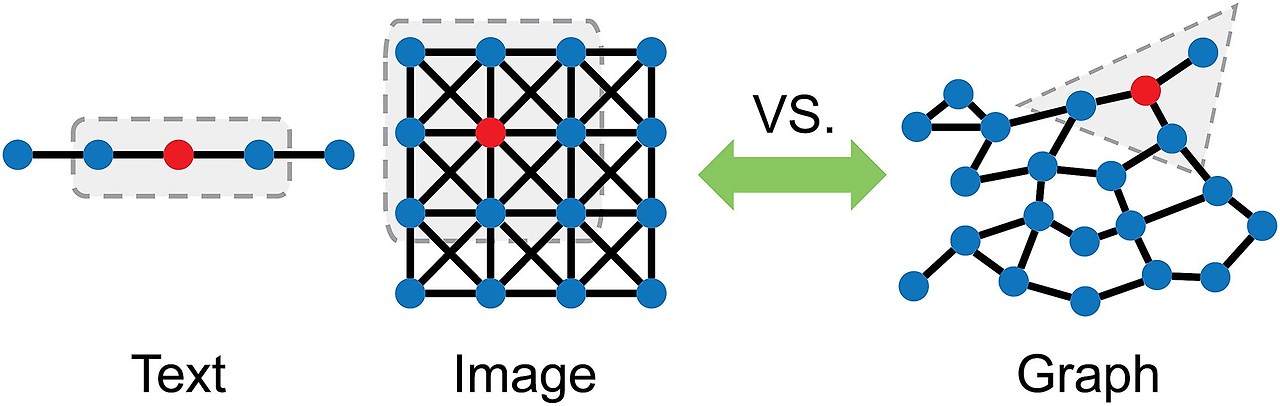
- 1D 데이터 (예: 텍스트)
- 순서가 정해져 있음 (linear structure)
- 예: "I love GNNs" → 순차적으로 처리 가능
- RNN, 1D CNN 등 사용
- 2D 데이터 (예: 이미지)
- 고정된 격자(grid) 구조
- CNN이 국소 영역(edge, corner 등)에서 특징 추출
- Graph 데이터
- 노드(node)와 엣지(edge)로 구성
- 이웃 노드 수나 연결 구조가 일정하지 않음
- 메시지 전달(message passing) 기반 학습 필요
- GNN은 이처럼 유동적이고 복잡한 연결 관계를 다룰 수 있는 프레임워크
텍스트(1D), 이미지(2D) 같은 전통적인 딥러닝 입력은 Euclidean 공간에 정의되어 있는 반면, 그래프(graph)는 Non-Euclidean 공간 구조에 존재한다는 점에서 본질적으로 다르다
GNN의 다양한 종류
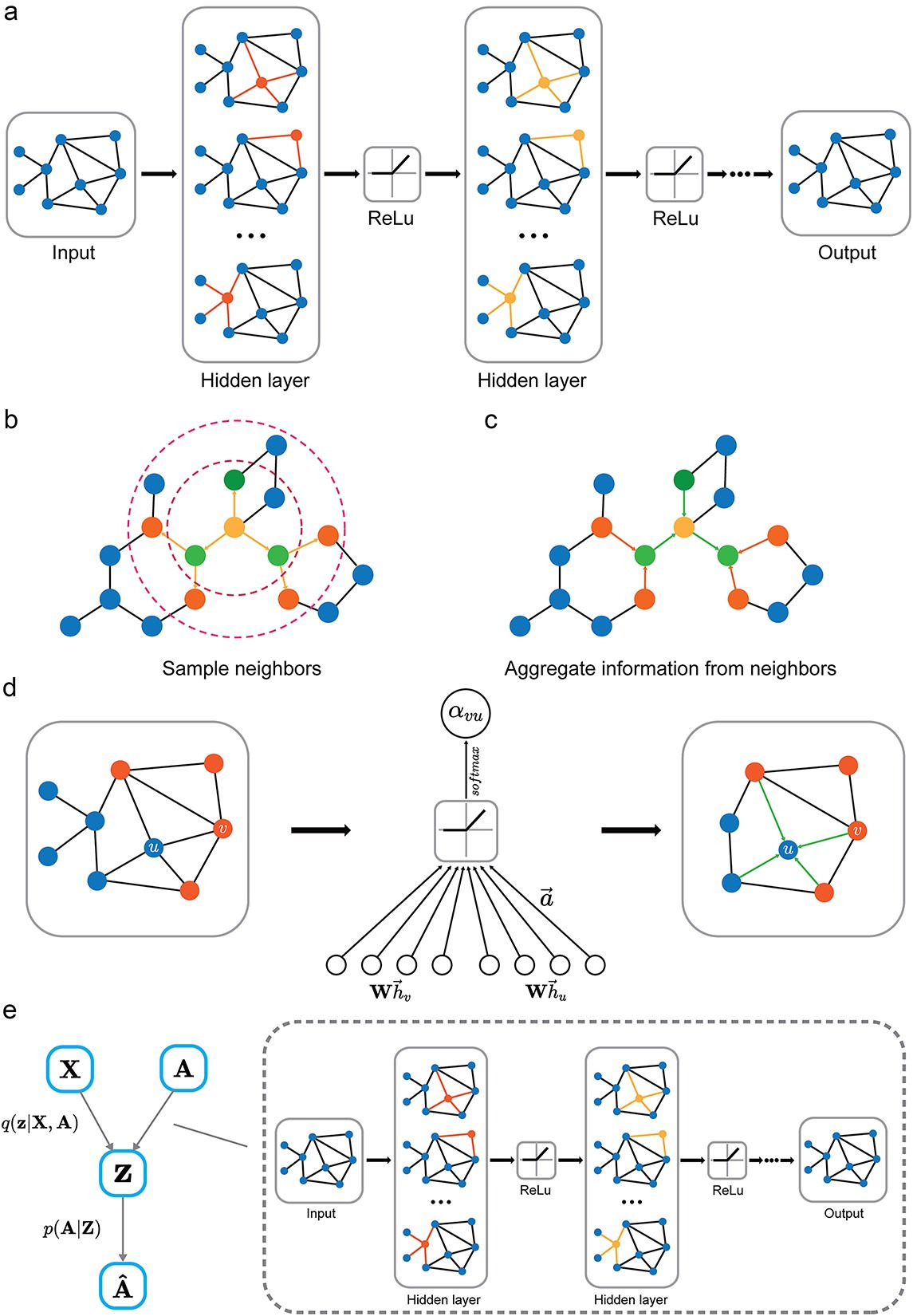
| GCN | 평균화된 이웃 정보 | 연산이 간단하지만 global context 부족 |
| GraphSAGE | 샘플링 기반 aggregation | large-scale graph에 적합 |
| GAT | attention 가중치 학습 | 이웃 중요도 반영 가능 |
| GTN | 그래프 + Transformer 스타일 | heterogeneous/multi-relational graph에 강력 |
| GAE/VGAE | autoencoder 구조 | 비지도 학습 (embedding, imputation) |
| MPNN | message passing generalization | 다양한 변형 가능 |
(a) GCN (Graph Convolutional Network)
- 전체 노드의 이웃 정보를 aggregate하고
- weight matrix와 활성화 함수(예: ReLU)를 거쳐
- node classification, graph classification 등의 task로 연결됨
- 일반적인 pipeline 구조:
- 입력 → GCN 레이어 → 활성화 → 풀링 → 출력
(b), (c) GraphSAGE
이웃이 너무 많아도 학습이 가능하도록 "neighbor sampling"을 도입한 GNN
- (b): 각 노드마다 고정된 수의 이웃을 샘플링 (e.g., 10개)
- (c): 샘플링된 이웃의 feature를 평균, LSTM, max 등 aggregation function으로 통합
- 장점: 대규모 그래프에서의 확장성 (scalability) 확보
(d) GAT (Graph Attention Network)
이웃 간 중요도를 학습된 attention score로 가중합
- 노드 v가 이웃 u로부터 정보를 받아올 때,→ 더 중요한 이웃에게 더 많은 영향을 부여
- 이웃마다 가중치를 학습함
- 구조:
- feature concatenation → attention layer → softmax → weighted sum
(e) VGAE (Variational Graph Autoencoder)
비지도(unsupervised) 방식으로 노드 임베딩을 학습하고
그래프 구조 자체를 복원하는 모델
- Encoder: GCN 등을 통해 노드를 latent vector로 임베딩
- Decoder: 두 노드 간 내적 또는 MLP로 edge 존재 여부 복원
- Loss: Variational loss (KL divergence + reconstruction loss)
활용: 네트워크 복원, 노드 임베딩, feature 추출 등에 적합
single cell omics에서 GNN의 활용


single cell omics data별 GNN 활용 툴
1. Epigenomics
1.1 Chromatin Accessibility (scATAC-seq)
Task: Cell type identification
요약:
- 세포 간 발현 유사도 또는 cross-omics (scRNA–scATAC) 유사성을 기반으로 hybrid graph 생성
- 다른 omics (scRNA-seq)의 레이블 정보를 활용해 cross-modal label transfer를 수행하거나
- 동일 omics 내에서 peak-level 또는 sequence-level feature를 활용하여 세포 임베딩을 학습
대표 모델:
- scGCN: scRNA-seq을 참조 데이터로 활용한 label transfer
- HyGAnno: gene-level과 peak-level feature 병합
- SANGO: peak sequence로부터 CNN + GTN 기반 예측
1.2 DNA Methylation (scDNAm)
Task: Methylation imputation
요약:
- cell과 CpG locus 간 known methylation 상태를 edge로 하는 bipartite graph 구성
- GCN으로 노드 임베딩을 학습하여 결측 메틸화 상태를 link prediction으로 예측
대표 모델:
- GraphCpG
1.3 Chromosome Conformation (scHi-C)
Task: Chromosomal interaction imputation, methylation prediction
요약:
- 각 세포를 chromosomal bins 간 상호작용으로 정의된 intra-cell contact graph로 모델링
- GNN과 transformer를 통해 전체 구조 또는 promoter-level interaction을 학습해 예측
대표 모델:
- HiC-SGL: 세포 그래프 상에서 interaction 복원
- scHiMe: GTN을 통해 3D genome 구조 기반 methylation 수준 예측
2. Transcriptomics (scRNA-seq)
2.1 Imputation
Task: Dropout된 발현값 복원
요약:
- 세포 간 Pearson correlation, PCA embedding 거리, 또는 kNN 기반의 cell–cell similarity graph 생성
- GNN 또는 attention을 사용해 이웃 세포로부터 누락된 발현값을 보완
대표 모델:
- GraphSCI: Pearson correlation + GCN + AE
- GNNImpute: kNN + attention 가중치
2.2 Dimensionality Reduction
Task: 저차원 임베딩 학습
요약:
- raw 발현값 또는 차원 축소된 feature로 계산한 cell–cell kNN graph 기반
- autoencoder 또는 GAE를 통해 노이즈를 제거하고 유의미한 임베딩 학습
대표 모델:
- scGNN: 반복적 클러스터링-재구성 기반 표현 학습
- scDHA-GNN, DR-GNN 등
2.3 Cell Type Identification
Task: 세포 클러스터링 및 분류
요약:
- 발현 유사도 기반 kNN 그래프 또는 feature correlation 기반 세포 간 그래프 구성
- kNN 기반 그래프에서 GCN 또는 self-supervised 학습을 통해
- 세포 간 구조를 반영한 유형 구분 수행
대표 모델:
- GraphSCC: dual self-supervised learning
- CellGNN, scGCL 등
2.4 Cell–Cell Communication
Task: 세포 간 상호작용 예측
요약:
- 세포 간 발현 correlation 또는 ligand–receptor DB 기반 binary 또는 weighted cell–cell graph 구성
- VGAE, GAT 등을 사용해 세포 간 신호 전달 관계를 예측
대표 모델:
- Graph-DiffVAE: VGAE 기반
- GraphComm: annotated ligand-receptor DB 활용
- PathFinder: GTN 기반 signaling path 분해
3. Spatial Transcriptomics (SRT)
3.1 Spatial Gene Expression Prediction
Task: 이미지로부터 유전자 발현 예측
요약:
- patch 간 Euclidean 거리 기반으로 spot-level kNN 그래프 구성
- 조직학 이미지로부터 patch feature 추출
- spatial transformer + GNN을 통해 공간적 gene expression 예측
대표 모델:
- Hist2ST
3.2 Dimensionality Reduction
Task: 공간 구조 반영 저차원 임베딩
요약:
- spot 위치의 spatial coordinate 기반 kNN 혹은 radius-based neighbor graph 생성
- GNN을 통해 공간적으로 구분되는 세포 표현 학습
대표 모델:
- spage2vec
- STAGATE, SpaGCN (일부 해당됨)
3.3 Spatial Domain Identification
Task: 조직 내 기능적 영역 정의
요약:
- spot 간 spatial 거리 + 발현 유사도 + 조직학 정보를 통합한 weighted graph 구성
- GCN 기반 클러스터링을 통해 공간 도메인 및 변이 유전자 추출
대표 모델:
- SpaGCN
- STAGATE, SEDR 등
3.4 Cell–Cell Communication
Task: 위치 기반 상호작용 예측
요약:
- 세포 위치 간 거리 기반 threshold 적용하여 neighbor graph 구성 (ligand-receptor 분석용)
- GCN을 통해 ligand-receptor 기반 상호작용 네트워크 예측
대표 모델:
- GCNG
4. Proteomics
Task: Clustering, classification, tissue 상태 분석
요약:
- 단백질 abundance 간 Pearson correlation 또는 이미지 기반 이웃 관계로 cell–cell graph 생성
- 또는 이미지 기반 spatial 정보 활용
- GCN을 통해 단백질 기반 세포 표현 및 면역 반응 예측
대표 모델:
- scPROTEIN: abundance 기반 cell graph
- SNOWFLAKE: 이미지 + proteomics 통합
5. Multi-omics Integration
5.1 Omics Alignment
Task: scRNA, scATAC, scDNAm 등 통합
요약:
- 유전자/peak/feature 간 known regulatory 관계 기반 feature–feature guidance graph 생성
- omics별 VAE로 표현 학습 후
- prior knowledge 기반 guidance graph로 정렬
- shared cell embedding 학습
대표 모델:
- GLUE
5.2 Rare Cell Population Detection
Task: 희귀 세포 탐지 및 조절 네트워크 추론
요약:
- 세포–유전자–peak 간 이종 관계를 edge로 하는 heterogeneous graph 구성
- 확률 기반 transformer 모델로 희귀 클러스터 식별 및 eGRN 추론
대표 모델:
- MarsGT
5.3 Biological Network Inference
Task: gene–cell, gene–gene, cell–cell 네트워크 추정
요약:
- gene–cell matrix로부터 gene–gene, cell–cell 간 correlation을 edge로 갖는 multiple graphs 구성
- VGAE 및 embedding을 통해 연관성 예측 및 pathway 활성 추정
대표 모델:
- scapGNN
5.4 Cell Type Deconvolution (ST 기반)
Task: spot 단위 세포 구성 비율 추정
요약:
- pseudo-ST와 실제 ST spot 간 공동 kNN 기반 spot–spot graph 구성
- semi-supervised GCN으로 spot 내 cell composition 추정
대표 모델:
- DSTG
앞으로의 방향성
- 더 생물학적으로 해석 가능한 모델
- 기존 지식(예: 유전자 네트워크, 단백질 상호작용(PPI, TF–target, ligand–receptor)등) 통합
- 파운데이션 모델(사전학습된 모델)과의 결합 가능성